
Сравнение первичных аминокислотных последовательностей фермента MAP3K7
Цель работы: сравнить первичные аминокислотные последовательности, используя базу данных UniProt, программу Clustal и программу Blast. Составить сравнительную таблицу в Excel, где сравнить данные полученные при использовании программы Blast и базы данных UniProt.
Для этого мы рассмотрим следующие организмы: ![]() Human- человек; Наиболее
похожие:
Human- человек; Наиболее
похожие:
![]() Mustela putorius - лесной
хорёк;
Mustela putorius - лесной
хорёк;
Оdobenus rosmarus - морж;
Ictydomys tridecemlineatus - суслик тринадцать полос;
Tursiops truncates - дельфин (Афалина); Lipotes vexillifer - китайский речной дельфин;
Sus scrofa - кабан;
Sheep - овца;
Goat - коза домашняя;
Rattus norvegicus - серая крыса;
Orycotolagus cuniculus - дикий кролик;
Mesocricetus auratus - сирийский хомячок;
Mus Musculus - домовая мышь;
Loxondata African - саванный слон;
Sarcophilius harrisii - тасманский дьявол(хищный сумчатый); Bos taurus - домашний бык; Наименее похожие:
![]() Myotis lucifugus - малая
бурая ночница (летучая мышь);
Myotis lucifugus - малая
бурая ночница (летучая мышь);
Cavia porcellus - морская свинка;
Taenipygia gutta - зебровая амадина (птица);
Austrofundulus limnaeus - рыба;
Salmo salar- атлантический лосось;
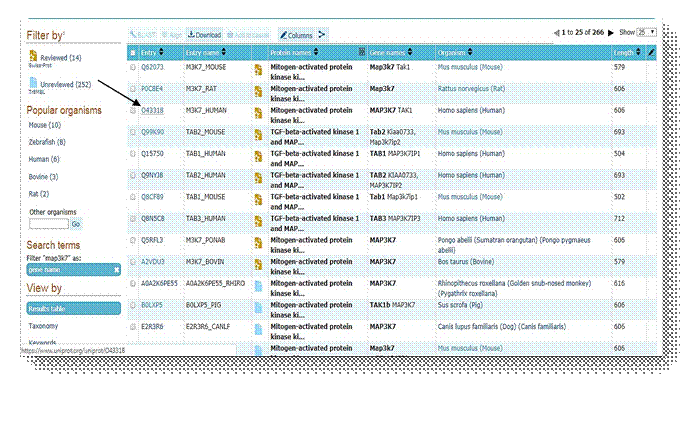
1) Поиск первичных аминокислотных последовательностей в системе UniProtKB.
 |
Далее выбираем из списка наиболее изученные белки.
Золотым значком со звездочкой отмечены полностью расшифрованные белки, а голубым значком – белки, расшифрованные не полностью (отсутствует информация о каком-либо сайте и т.д.)
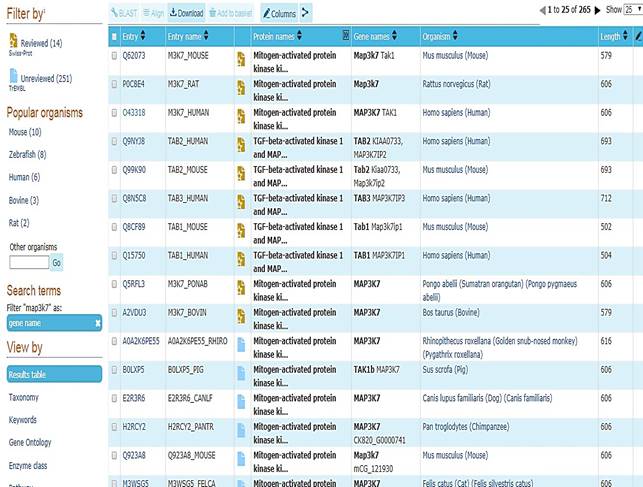
Показан результат поиска исследуемого фермента MASPK7. В таблице указано: номер, название рецептора, название белка, название генов, кодирующих белок, организм и длина последовательности.
Выбираем объекты, необходимые для исследования, и скачиваем их FASTA. FASTA - это документ, в котором содержится аминокислотная последовательность белка. Сохраняем ее в формате txt.

2) Попарное сравнение аминокислотных последовательностей MASPK7 человека с другими организмами в программе ClustalX2.
Сравниваем аминокислотные последовательности в программе Clustal. Для этого открываем программу Clustal и добавляем нужные последовательности командой File, Load Sequences. Выбираем ранее скачанные FASTA.
Затем загружаем белок человека:
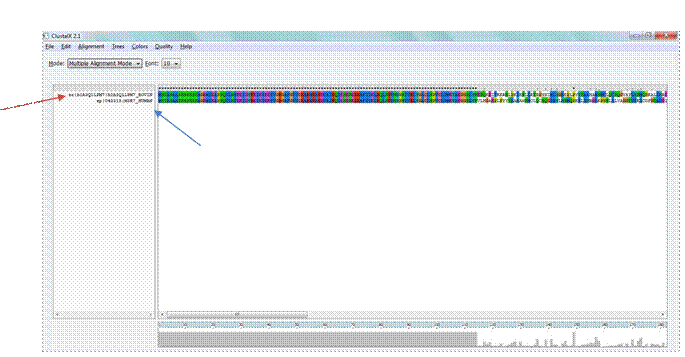
На данной картинке сравниваются рецепторы человека и быка. Над последовательностями отображаются значки звездочка (*), двоеточие (:) и точка (.):
(*) - идентичные аминокислотные остатки или нуклеотиды;
(:) - консервативные замены;
(.) – полуконсервативные замены.
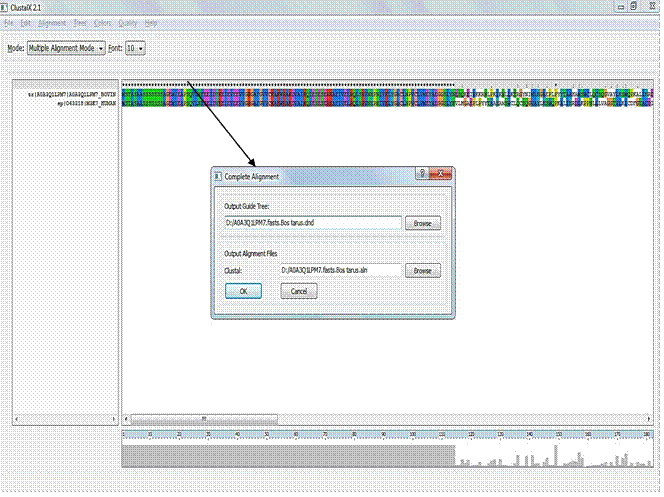
Затем выполняем выравнивания. Для этого выбираем вкладку Alignment Do Complete Alignment. Получаем то, что представлено выше на рисунке.
Выравнивание делаем для каждого варианта.
3) Попарное сравнение аминокислотных последовательностей MASPK7 человека с другими организмами в системе BLAST.
Заходим на сайт программы: http://blast.ncbi.nlm.nih.gov/Blast.cgi
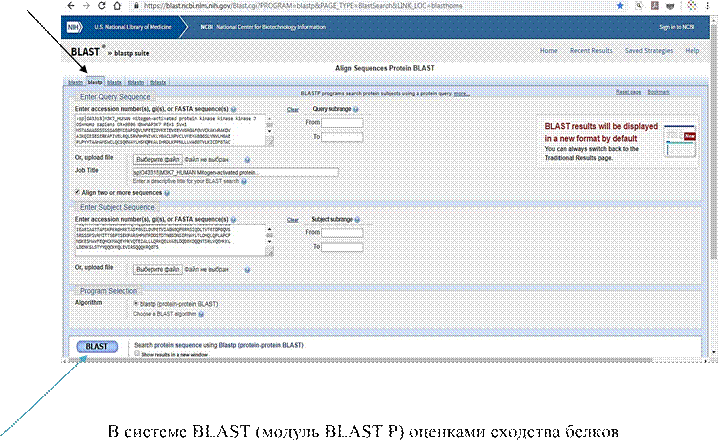
В программе Blast выбираем blastp (protein blast). Выбираем ранее скачанные FASTA в базе данных UniProt.
Сначала загружаем человека, затем животного. Помечаем галочкой Align two or more sequences, чтобы сравнивать последовательности двух объектов. Нажимаем команду BLAST.
служили 5 показателей:
![]() Max score - Максимальная оценка выравнивания из базы
данных последовательностей;
Max score - Максимальная оценка выравнивания из базы
данных последовательностей;
![]() Total score - Общий балл
от всех выравниваний;
Total score - Общий балл
от всех выравниваний;
Query cover - процент запросов, охватывающих выравнивание последовательностей базы данных;
![]() Значение E –доверительная вероятность;
Значение E –доверительная вероятность;
Max ident - самый высокий процент идентичности.
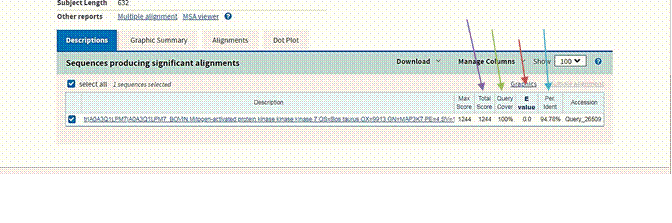
Находим значения Positives.
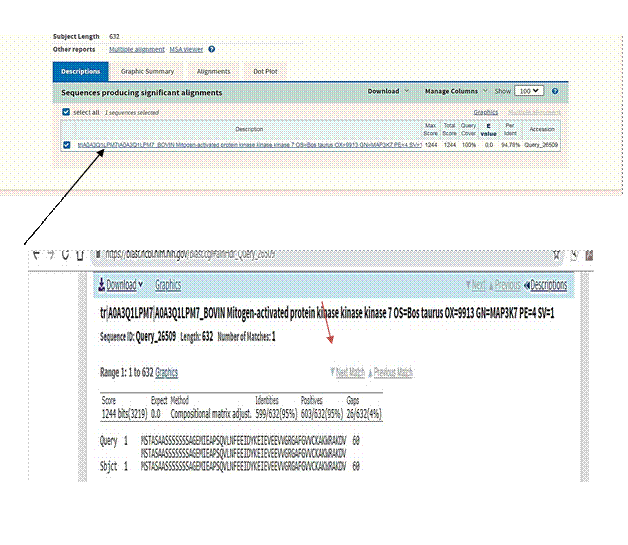
4) Проделав все действия в Blast, и получив нужные нам значения, заполняем таблицу в Excel
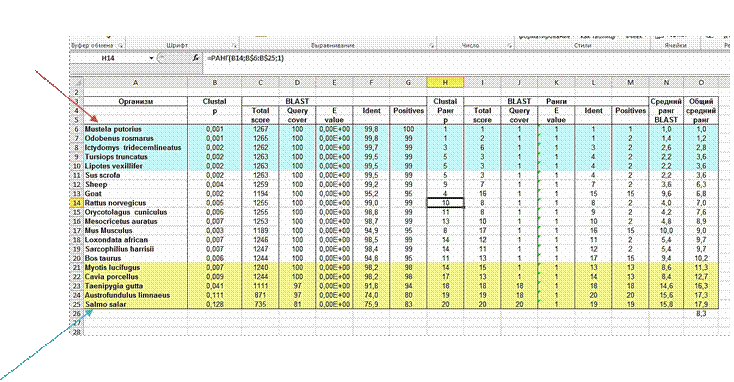
Вывод: Мы использовали биотехнологии чтобы сравнить аминокислотные последовательности MAP3K7 в системах ClustalX2 и BLAST. Наиболее схожим по первичной структуре с белком человека оказался белок Mustela putorius - лесной хорёк. Таким образом, при исследовании ингибитора MAP3K7, наиболее рационально использовать это животное. Проводим ранжирование, что позволяет нам осуществить обобщенный анализ разных по характеру данных (проводим консенсусный подход). Вычислив средний ранг, самый непохожий организм –
Salmo salar - атлантический лосось.
Цель: найти модели белка человека и животного и сравнить их.
Построение структурных моделей в биоинформатике предполагает изображение геометрии объекта, его поверхности, среды объекта, цвета.
При построении 3D моделей MAP3K7 использовали банк UniProt – для поиска последовательностей белка и обнаружения 3D моделей.
 |
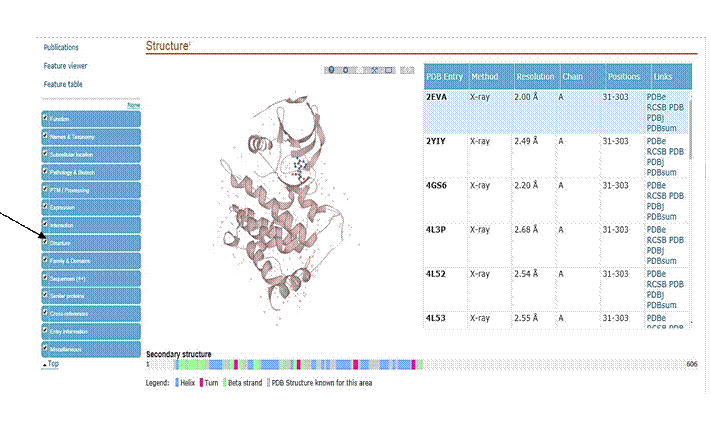 |
Выбираем наиболее подходящую модель по определенным критериям:
1. Рентгеноструктурная модель (X-ray);
2. Разрешение не более 3 ангстрем (около 2-х);
3. Цепочка должна быть одна;
4. Белок должен быть как можно более длинным.
Под эти условия подходит 1 модель:
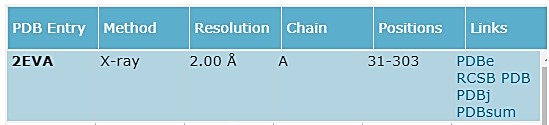
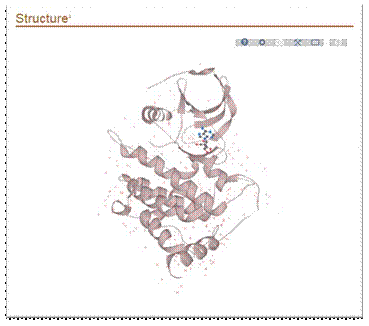
Model Human
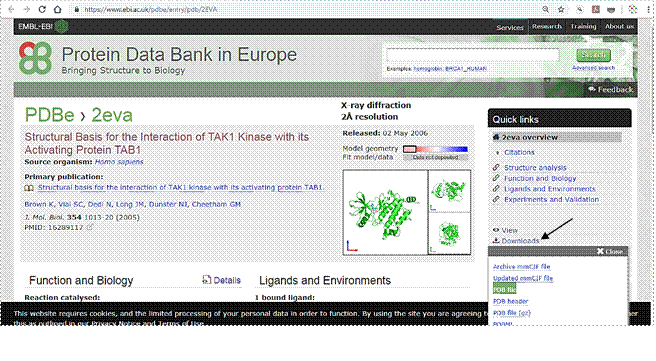
Переходим на страничку Protein Data Bank, и нажимаем Downloads (формат PDB file):
Открываем программу Chem 3D (с загруженным ранее белком человека):
 |
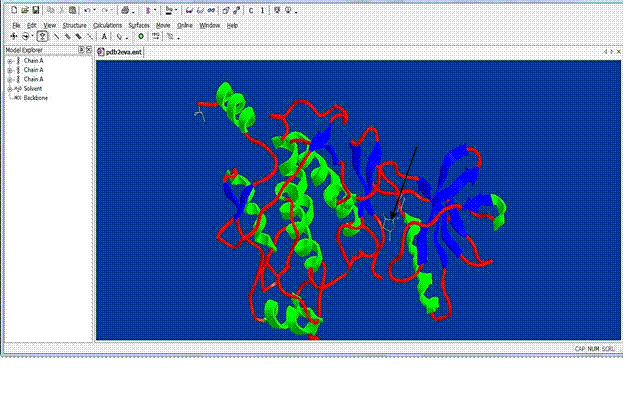
В центре мы наблюдаем лиганд, который связывается со спецефическим сайтом.
Переходим на страничку Protein Data Bank, смотрим какие лиганды присущи данному белку:

Выбираем наиболее подходящий лиганд:
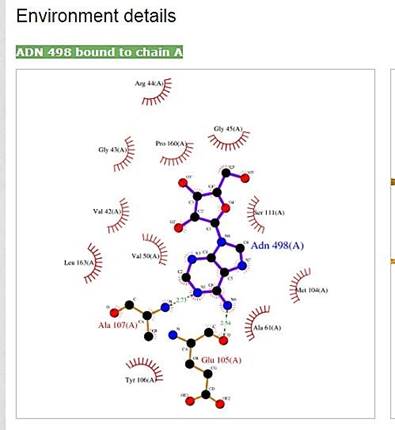
Выписываем все аминокислоты:
Находим наш лиганд:
(Display Mode; Space Filing)
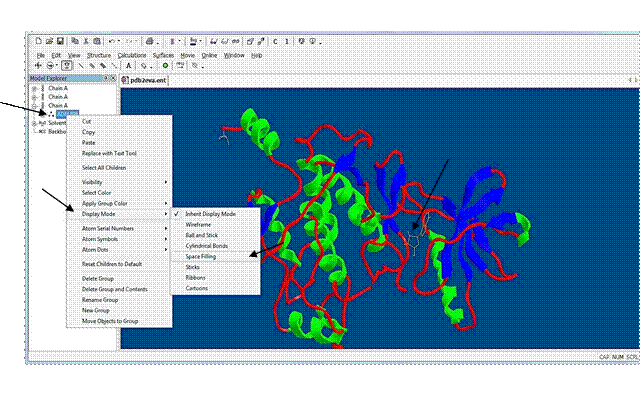
Лиганд превращается в шариковую модель:
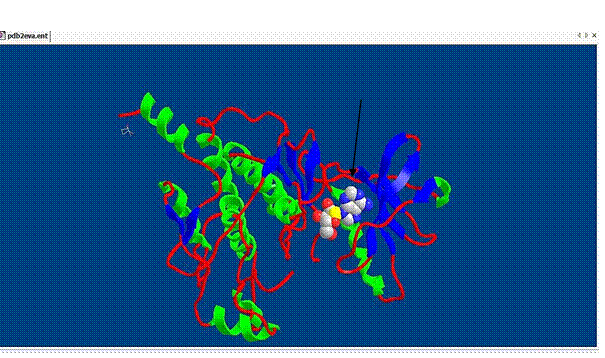
В программе Chem 3D Ultra белок можно рассматривать с разных сторон, выбирая нужный градус.
Также можно осуществлять приближение, т.е. увеличение модели, для удобства изучения структуры, воспользовавшись для этого значком на панели управления, изображенном на рисунке выше.
Зеленым цветом обозначаются альфа-спирали белка, а красным – неструктурированные фрагменты. Также имеются синие стрелки, помогающие легко посчитать количество бета-листов.
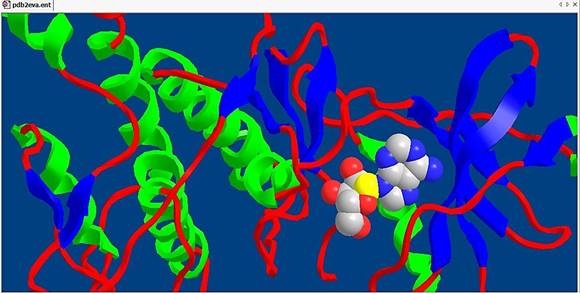
Обозначаем на данной модели аминокислоты (белый цвет):
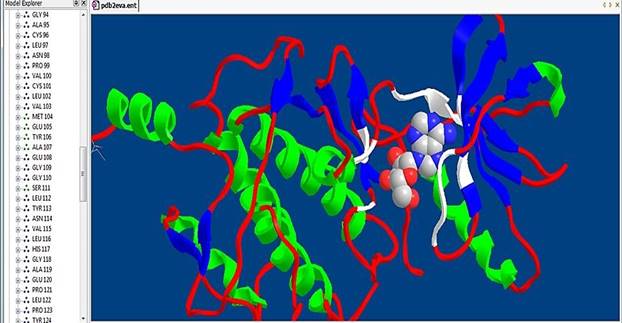
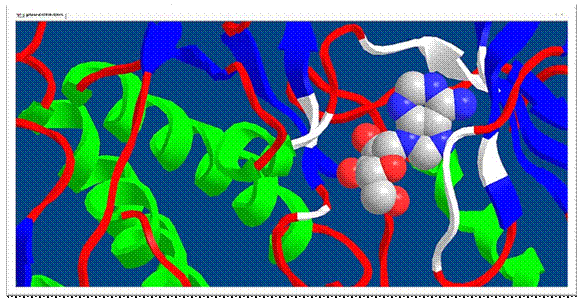
Сохраняем файл с данной окрашенной моделью.
![]()
Анализируем модель мышки
Переходим в колонку Structure, затем на сайт ModBase:
По критерим выбираем наиболее подходящию модель:

 |
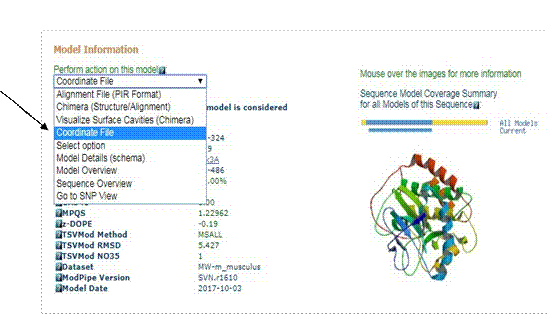
Открываем программу Chem 3D (с данным файлом):
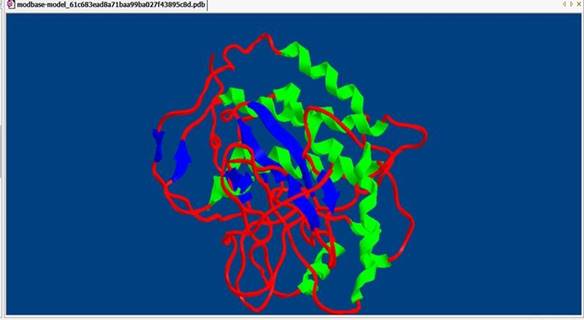
Model Mouse
Находим аминокислоты:
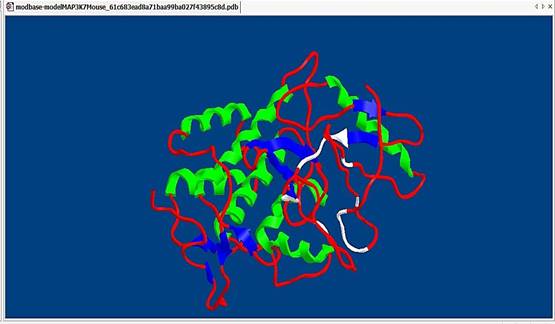
Сохраняем окрашенный файл( мышь):
![]()
Должны получиться файлы таких форматов:
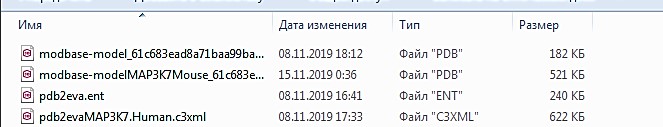
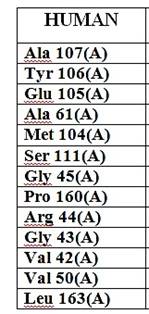
Сравнивание белка человека и мыши:
|
HUMAN |
MOUSE |
Cходство |
|
Ala 107(A) |
Ala 107(A) |
+ |
|
Tyr 106(A) |
Tyr 106(A) |
+ |
|
Glu 105(A) |
Glu 105(A) |
+ |
|
Ala 61(A) |
Ala 61(A) |
+ |
|
Met 104(A) |
Met 104(A) |
+ |
|
Ser 111(A) |
Ser 111(A) |
+ |
|
Gly 45(A) |
Gly 45(A) |
+ |
|
Pro 160(A) |
Pro 160(A) |
+ |
|
Arg 44(A) |
Arg 44(A) |
+ |
|
Gly 43(A) |
Gly 43(A) |
+ |
|
Val 42(A) |
Val 42(A) |
+ |
|
Val 50(A) |
Val 50(A) |
+ |
|
Leu 163(A) |
Leu 163(A) |
+ |
Вывод: Получили 3D модели MAP3K7 для человека, и убедились в том, что структурные модели позволяют определить функционально значимые участки в аминокислотных последовательностях. Проанализировали визуально эти модели. Связывающие аминокислоты человека и мыши - идентичны.
Общий вывод: Выполнено сравнение сходства 20 животных с белком человека. Установленно, что наиболее похожих животных выделяют 5, из них наиболие хорошим является Mustela putorius (лесной хорек). Таким образом, из мяса хорька можно получать чистый белок для проведение эксперементов in vitro или ферментативного катализа. Кроме этого, с целью уточнения результатов, проведено исследование функциональных участков белка человека с такими же участками - каталитическим сайтом. Связывающие аминокислоты в сайте человеческого белка и белка мыши являются идентичными, поэтому мышь можно использовать в качестве лабораторного животного для изучения данного белка.
Материалы на данной страницы взяты из открытых источников либо размещены пользователем в соответствии с договором-офертой сайта. Вы можете сообщить о нарушении.